近日,永利集团88304于彬教授团队在生物信息学研究领域取得重要进展,相关成果以“Exploring the Latent Information in Spatial Transcriptomics Data via Multi-View Graph Convolutional Network Based on Implicit Contrastive Learning”为题发表在综合领域1区顶级期刊Advanced Science (中科院一区TOP期刊,影响因子14.3)。

随着人工智能和生物信息学的快速发展,空间转录组学数据挖掘研究已成为生物信息学研究的热点前沿。它聚焦于细胞之间的相互作用、组织与器官微环境以及不同空间区域内基因表达差异等关键问题,这些问题的深入探索对理解复杂生物系统至关重要。传统转录组学技术,如scRNA-seq,虽能定量分析细胞或组织的整体转录本,但在处理组织样本时会丢失空间位置信息,难以全面解析生物系统。利用人工智能强大的数据分析和处理能力,结合海量的生物信息大数据,空间转录组学能够更好地将细胞的基因表达与空间位置信息相结合,在保留组织结构的同时,精确获取并定位每个细胞或组织区域的基因表达数据,为生命科学研究带来新的突破。
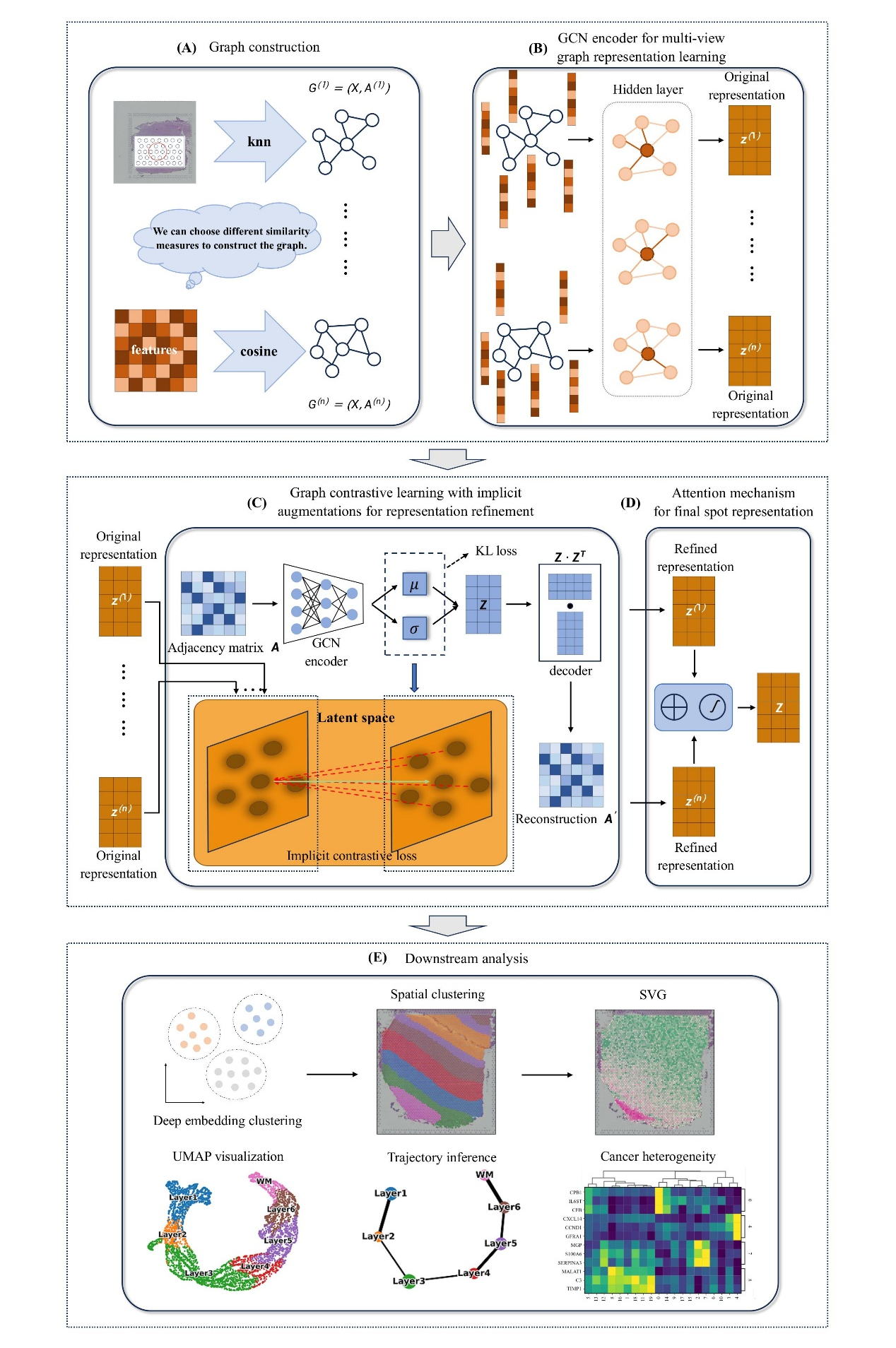
研究团队提出一种基于隐式对比学习的多视图图卷积网络框架,称为 STMIGCL。STMIGCL 借助空间转录组数据中的基因表达谱以及空间位置信息,运用不同的方法构建多个邻居图,依靠不同视图间的协同学习从数据中提取更为丰富且有效的信息。运用GCN学习每个视图的低维表示,实现图结构与基因表达特征的有效融合。通过将多视图学习与对比学习相结合,STMIGCL可充分利用GCN的独特优势深入挖掘每个视图中的潜在信息,还可以有效整合并利用不同视图之间的互补优势,获取更全面、丰富的信息。进一步利用基于隐式增强的对比学习方法,STMIGCL 显著增强表示的判别能力,优化聚类性能。为了捕捉不同视图嵌入的重要性,STMIGCL 运用注意力机制,自适应融合不同视图的潜在表示,得到最终的斑点表示。通过在多个不同技术平台获得的数据集中进行实验的结果表明,STMIGCL显著提升空间域的识别精度,在轨迹推断、空间可变基因识别等任务中优于现有基线方法。另外,通过在人类乳腺癌数据集上的实验分析结果表明,STMIGCL能够从更精细的层次剖析癌组织的异质性,为制定靶向治疗策略提供理论依据。
2022级硕士研究生任胜是第一作者,永利集团88304于彬教授和沙特阿卜杜拉国王科技大学高欣教授是该论文的通讯作者,本研究得到国家自然科学基金、山东省自然科学基金等项目资助。
Sheng Ren, Xingyu Liao, Farong Liu, Jie Li, Xin Gao*, Bin Yu*. Exploring the Latent Information in Spatial Transcriptomics Data via Multi-View Graph Convolutional Network Based on Implicit Contrastive Learning. Advanced Science, 2025, 2413545.
原文链接:https://doi.org/10.1002/advs.202413545.